6.11 组学联合分析 EMP_multi_analysis
模块EMP_multi_analysis旨在通过统计分析方法将多个具有统计学意义的p值整合到一个综合的p值中。通过参数method指定联合分析的方法,包括:feature、diff_feature_enrich和same_feature_enrich。
6.11.1 融合不同研究队列中相同特征的统计结果
不同的研究队列经常会针对同一个研究目标进行分析,其研究结果可能不一致。在微生物研究中,这种情况尤为普遍。为了应对这种情况,我们可以采用融合p值的方法,将多个统计学结果合并为一个综合结果。
🏷️示例:
从MAE对象中提取两个区域的微生物物种注释数据物种,并进行差异性分析。
k1 <- MAE |>
EMP_assay_extract('taxonomy') |>
EMP_filter(Region == 'Paris') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group)
k2 <- MAE |>
EMP_assay_extract('taxonomy') |>
EMP_filter(Region == 'Guangzhou') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group)
使用融合p值的方法,融合两项差异性分析的p值结果。
注意:
①参数
②参数
①参数
combineMethod提供三种融合P的方法:fisher,edgington 和 stouffer。②参数
action='get'提取差异性分析结果。
(k1+k2) |>
EMP_multi_analysis(method = 'feature',combineMethod='edgington',
p.adjust = 'BH')

6.11.2 融合不同研究队列中相同特征的KEGG富集结果
本模块支持合并不同队列中相同特征的KEGG富集结果。
🏷️示例:
从MAE对象中提取两个区域的微生物功能基因的KO注释数据,并进行差异性分析和富集分析。
k1 <- MAE |>
EMP_assay_extract('geno_ko') |>
EMP_filter(Region == 'Paris') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_enrich_analysis(pvalue < 0.05,keyType = 'ko')
k2 <- MAE |>
EMP_assay_extract('geno_ko') |>
EMP_filter(Region == 'Guangzhou') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_enrich_analysis(pvalue < 0.05,keyType = 'ko')
对于相同特征的的富集分析结果,本模块有三种方法可以进行融合:enchier, ActivePathways和mitch。
注意:
①当使用
②
①当使用
enchier方法时,采用的是p值融合算法。通过调整combineMethod参数,可以决定融合p值发生在特征水平还是富集分析结果水平。②
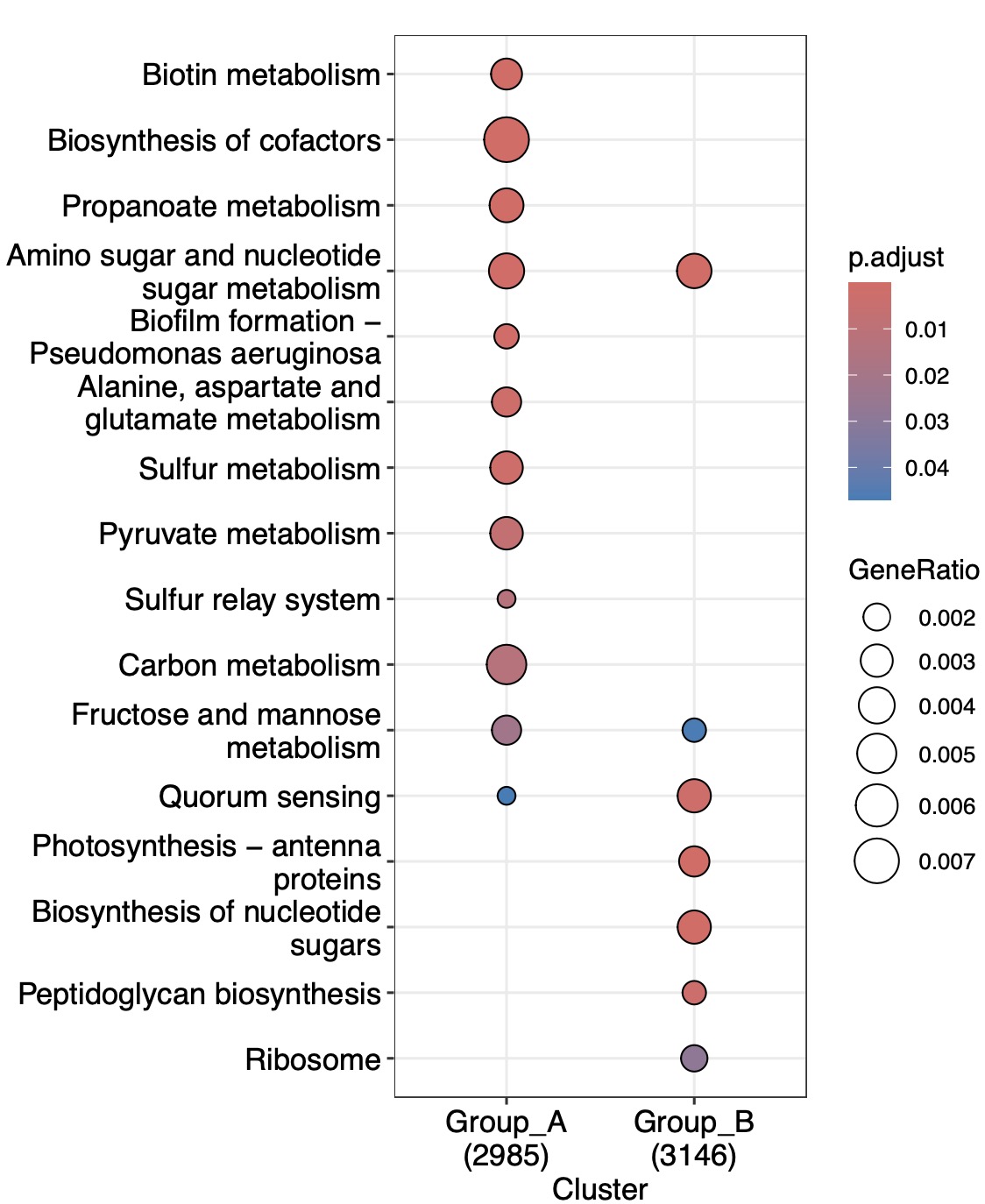
ActivePathways和mitch算法则依赖于已经发表的两个组学融合分析包。(k1+k2) |>
EMP_multi_analysis(method = 'same_feature_enrich',
keyType = 'ko',combineFun='ActivePathways')|>
EMP_enrich_dotplot()
6.11.3 融合相同队列不同特征的KEGG富集结果
在同一个队列中,有时候会同时进行功能基因和代谢组学的测序分析,因此会出现两套KEGG富集分析的结果,且这两套结果通常不完全一致。在此种情况下,单纯使用韦恩图求交并集不能很好地揭示组学之间的富集情况。为了应对这种情况,本模块利用算法将不同组学中不同特征的KEGG富集结果融合在一起。
🏷️示例:
从MAE对象中提取KO注释结果和代谢组学结果,并分别完成KEGG富集分析。
k1 <- MAE |>
EMP_assay_extract('geno_ko') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_enrich_analysis(pvalue < 0.05,keyType = 'ko')
k2 <- MAE |>
EMP_collapse(experiment = 'untarget_metabol',na_string=c('NA','null','','-'),
estimate_group = 'MS2kegg',method = 'sum',collapse_by = 'row') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_enrich_analysis(pvalue < 0.05,keyType = 'cpd')
使用+符号将组学联合进行富集分析。
(k1+k2) |>
EMP_multi_analysis(method = 'diff_feature_enrich') |>
EMP_enrich_dotplot()
